mirbase数据库怎么引物设计
发布时间:2025-05-22 18:32:18 发布人:远客网络
一、mirbase数据库怎么引物设计
1、miRNA可以设计茎环引物反转录,用探针法或者染料法检测。这个引物设计都是固定格式的
2、miRNA引物设计(茎环法+染料法检测)
3、设计方法:通用茎环后端加miRNA后面6个碱基的反向互补序列为反转录引物,正向引物为miRNA去除后面6个碱基的序列,反向引物在茎环上。
4、GTCGTATCCAGTGCGTGTCGTGGAGTCGGCAATTGCACTGGATACGAC
5、>mmu-miR-99b-5p MIMAT0000132 Mus musculus miR-99b-5p
6、mmu-miR-99b-5p反转录颈环引物为:
7、GTCGTATCCAGTGCGTGTCGTGGAGTCGGCAATTGCACTGGATACGACCGCAAG
8、定量PCR反向引物为:TGTCGTGGAGTCGGC
9、1定量PCR反向引物和正向引物的TM值不要相差太大,反向引物TM已经确定,如果正向引物TM较低,则在5`端添加几个碱基(如:G)。
10、2染料法的检测可能会出现引物二聚体和非特异性扩增,可以使用探针法检测,探针法和染料法相似,只是茎环上不仅有一个反向引物结合位点,还有一个探针结合位点。
二、分析miRNA的数据库都有哪些
一个高通量实验数据CLIP-Seq(或称为HITS-CLIP,PAR-CLIP,iCLIP)和mRNA降解组测序数据支持的microRNA靶标数据库,包含了miRNA-mRNA,miRNA-lncRNA,miRNA-circRNA,miRNA-ceRNA和RNA-protein等的调控关系。整合和构建多个流行的靶标预测软件的交集和调控关系。最新版本发布时间:2013年11月。
众所周知的microRNA基因注释数据库。目前miRBase只提供了microRNA的靶标的预测软件的链接(如:PicTar)。最新版本发布时间:2010年9月。
整合CLIP-Seq和ChIP-Seq的数据探讨microRNA的转录和转录后调控,构建转录因子->microRNA->靶标的调控网络。最新版本发布时间:2012年11月。
一个收集已被实验验证的microRNA靶标数据库。最新版本发布时间:2009年1月。
一个整合的microRNA靶标数据库。整合多个靶标预测软件的调控关系。最新版本发布时间:2010年11月。
基于靶mRNA序列的进化保守等特征搜寻动物的microRNA靶基因。是预测microRNA靶标假阳性率较低的软件。而且是microRNA领域大牛Bartel实验室开发的。最新版本发布时间:2009年4月。
基于microRNA或microRNA靶标联合作用等特征开发的搜寻动物的microRNA靶基因。假阳性率也较低。是microRNA领域大牛Rajewsky实验室开发的。最新版本发布时间:2007年3月。
基于靶位点的可接性和自由能预测microRNA的靶标。是著名的生物信息学家Segal实验室开发的。最新版本发布时间:2008年8月。
基于序列特征预测microRNA的结合位点。是几个流行的microRNA靶标预测软件的其中一个。IBM公司的研究团队开发的。最新版本发布时间:2007年。
是著名的MemorialSloan-Kettering癌症研究中心的研究人员开发的软件和数据库。miRanda的最新版本又叫mirSVR。最新版本发布时间:2010年8月。
EMBL-EBI的Enright实验室开发的microRNA靶标数据库。最新版本发布时间:2010年8月。
整合实验证实的microRNA靶标的数据库。最新版本发布时间:2010年10月。
整合microRNA表达、靶标和疾病相关信息的数据库。最新版本发布时间:2010年11月。
动物的microRNA基因及其靶标的数据库。最新版本发布时间:2008年1月。
动物microRNA靶标预测和功能注释数据库。最新版本发布时间:2010年8月。
一个基于miRNA-target配对自由能预测microRNA的靶标。最新版本发布时间:2011年6月。
microRNA基因和microRNA靶标数据库。最新版本发布时间:2007年1月。
三、快速查找miRNA成熟体名称--miRBase数据库中ID转换的两种方法
miRBase数据库(mirbase.org/)作为microRNA序列和注释的主要公共存储库和在线资源,提供最新版本(v22)的38589个条目,覆盖271种生物体的hairpin precursor microRNAs。数据库允许通过hairpin precursor和成熟序列进行搜索和浏览,同时支持基于名称、关键字、引用和注释对条目进行检索。所有序列和注释数据均可下载。
在进行数据分析时,若需将miRBase数据库中的官方ID转换为miRNA基因ID,可以采用以下两种方法。
方法1:通过miRBase数据库下载对应文件
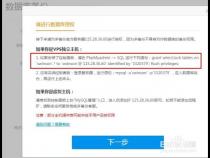
步骤1:访问miRBase数据库官网(mirbase.org/)。
步骤2:进入下载界面(mirbase.org/ftp/CURRENT...)。
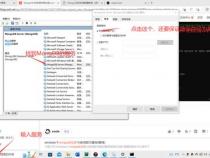
步骤3:下载miRNA.xls.zip文件。
步骤4:解压文件后,即可获取到对应的ID信息。
miRBaseVersions.db是一个注释包,内含22个miRBase发布版本的成熟miRNA名称。此包可用于快速查找成熟miRNA名称。通过使用包中的四个函数(Columns、Keytypes、Keys、Select)来完成ID匹配。首先,安装R包:
使用命令`if(!requireNamespace("BiocManager", quietly= TRUE)) install.packages("BiocManager")`安装BiocManager,再使用`BiocManager::install("miRBaseVersions.db")`安装miRBaseVersions.db包。
Keytypes:接收可以检索数据的表名列表。
Columns:检索用于最终输出的变量类型信息。
Keys:返回指定键类型的所有可行键。例如,检索miRBase版本6.0的所有可能键。
Select:提取数据,输入值为从其他三个函数(键、列和键类型)接收的输出。例如,提取成熟体'MIMAT0000092'的所有信息。
对于具体使用方法和示例,可参考生物计算平台bioconductor.org/packag...
通过以上两种方法,您可以实现从miRBase数据库官方ID到miRNA基因ID的转换,从而进行进一步的数据分析。