数据库推荐丨肿瘤数据库,可不只有TCGA、GEO!
发布时间:2025-05-22 10:50:47 发布人:远客网络
一、数据库推荐丨肿瘤数据库,可不只有TCGA、GEO!
1、在研究肿瘤数据库时,广为人知的TCGA和GEO确实有其价值,但本文将为您揭示一些鲜为人知但同样实用且持续更新的数据库资源。这些数据库按照综合、基因组和转录组类别划分,帮助您更全面地探索癌症相关数据。
2、TCGA(cancergenome.nih.gov/),曾有我们GCBI知识库的教程视频,涵盖了数据下载、整合和应用,详情可通过留言获取链接。
3、COSMIC(cancer.sanger.ac.uk/cos...),是全球最大的肿瘤体细胞突变资源,提供基因组变异、表达等信息。
4、UCSC Cancer Genomics Browser(genome.ucsc.edu/index.h...),可视化分析癌症基因组学和临床数据,包含大量样本数据。
5、ArrayMap(arraymap.org/),提供预处理的基因组芯片数据和CNA图谱。
6、Cancer Hotspots(cancerhotspots.org/#...),关注复发突变热点,来自大规模基因组学数据。
7、OncoKB(oncokb.org/#...),MSK维护的精准肿瘤学知识库,包含治疗指导和基因改变信息。
8、最后是肿瘤转录组数据库,例如:
9、ArrayExpress(ebi.ac.uk/arrayexpress/),EMBL-EBI的基因组学实验数据仓库。
10、Oncomine(oncomine.org/),大型肿瘤基因芯片数据库,分析基因表达数据。
11、以上数据库各有其特色,可根据研究需求选择使用。
二、如何从GEO数据库下载数据
1、TCGA及GEO数据库是生信分析的两大主要数据来源,其中GEO数据库提供大量的临床与实验数据。本文旨在介绍如何从GEO数据库下载数据,主要分为两个示例:基因表达数据集与含有样本生存信息的临床特征数据集的下载方法。
2、下载基因表达数据集的步骤如下:首先进入GEO数据库首页,选择Datasets子数据库进行检索。输入关键词,如“lung cancer or LCA”,并进行筛选,选择数据集类型为“Series”,数据类型为“基因表达谱”,以及物种来源为“人类”。完成筛选后,根据研究标题、概述、平台注释、样本数量及数据集ID进行进一步筛选,以确保目标数据集的准确性。通过点击数据集标题进入详情页,确认数据集编号、介绍、检测平台及样本信息,最后点击下载按钮,下载表达矩阵“series matrix”文件和GPL平台注释文件。下载后,将平台注释文件与表达矩阵文件进行匹配,替换基因ID为基因名,得到基因表达谱。
3、下载含有样本生存信息的临床特征数据集的步骤与下载基因表达数据集类似,但需要在检索时加入关键词,如“OS”、“survival”、“Time”等,以获取更符合需求的数据集。在浏览实验样本数据时,确保样本临床特征信息中包含生存时间相关的数据。下载临床资料数据时,提取包含年龄、生存时间等信息的样本数据,并与基因表达谱文件合并,准备进行生存分析。
4、本文通过示例详细介绍了如何从GEO数据库下载所需的基因表达数据集与临床特征数据集,以支持后续的生信分析工作。通过上述步骤,研究人员能够有效地获取并处理数据,为研究提供坚实的基础。
三、seer数据库编码注释
1、seer数据库编码注释:seer数据库主要是临床特征的数据库,如果要找免疫组化及基因检测结果,上TCGA或者GEO数据库。
2、出现此现象时,数据库是正常的(做过一个实验:新建了一个网站,程序与运行网站相同,连接的数据库也与运行网站连接的数据库相同。当运行网站出现连不上数据库现象时,运行这个新网站,结果正常);出现此现象时,IIS也是正常的,因为网站其他不调用数据库的页面是可以打开的。
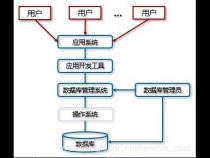
3、所谓的分布式数据库技术,就是结合了数据库技术与分布式技术的一种结合。具体指的是把那些在地理意义上分散开的各个数据库节点,但在计算机系统逻辑上又是属于同一个系统的数据结合起来的一种数据库技术。既有着数据库间的协调性也有着数据的分布性。
4、这个系统并不注重系统的集中控制,而是注重每个数据库节点的自治性,此外为了让程序员能够在编写程序时可以减轻工作量以及系统出错的可能性,一般都是完全不考虑数据的分布情况,这样的结果就使得系统数据的分布情况一直保持着透明性。