如何预测miRNA靶基因(miRWalk2.0数据库)
发布时间:2025-05-22 09:37:22 发布人:远客网络
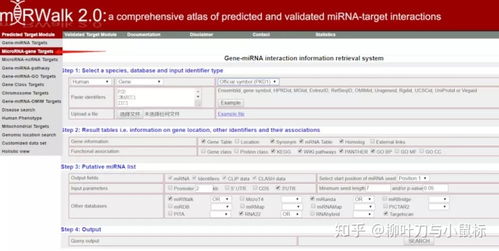
一、如何预测miRNA靶基因(miRWalk2.0数据库)
miRWalk2.0是miRWalk数据库的升级版,专为研究者提供全面的miRNA-mRNA相互作用预测和实验验证信息。该数据库不仅记录基因序列中的miRNA结合位点,还与12个知名数据库整合,包含多种类型的预测数据集。这些数据集涵盖基因的promoter、CDS、5’-和3’-UTR区域,同时整合了自动文本挖掘收集的实验验证信息及来源数据库提供的内容。
使用miRWalk2.0数据库时,研究者可通过输入不同基因ID(如基因symbol、EntrezID、EnsemblID或RefseqID)来预测miRNA-mRNA相互作用。根据基因类型,用户可选择查询5UTR、CDS、3UTR区域,通常miRNA靶位点主要在3UTR,因此直接下载3UTR数据即可。点击下载按钮即可获取相关数据。
针对miRNA查询,研究者只需输入miRBase给出的miRNA ID或来自其他数据库的miRNA编号,接受短名称或家族miRNA信息。完成查询后,用户可选择下载预测结果,文件内容与网页显示一致。
下载结果后,研究者可通过特定符号解读:红色箭头表示miRNA信息,橙色箭头表示mRNA信息,蓝色箭头代表使用的数据库,绿色箭头表示有意义的数据库总数(通常从高到低排序)。用户可以上传miRNA或mRNA列表,支持通过NCBI查询或输入gene symbol和EntrezID,但需注意单次输入基因ID或symbol数量限制,以确保数据准确。
miRWalk2.0不仅简化了miRNA-mRNA相互作用研究的流程,还提供了多维度的数据分析,助力科研人员深入探索miRNA生物学功能。通过合理利用该数据库提供的资源,研究者可以更高效地预测和验证miRNA靶基因,推动相关领域研究的进展。
二、miRwalk:搞定miRNA靶点预测及验证的宝藏网站
miRWalk数据库是一个功能全面的miRNA靶基因预测和验证工具,覆盖了人类、小鼠、大鼠、狗和牛等多种物种的基因信息。该数据库汇集了来自12个预测程序的miRNA结合位点信息,如DIANA-microTv4.0、DIANA-microT-CDS、miRanda-rel2010等,且定期更新,最新版本为V3。miRWalk支持双向预测模式,用户可以根据miRNA或基因进行预测。输入数据采用Gene symbols、Entrez Id或Ensembl-ID等形式,而miRNA识别支持mirBase ID、编号及家族名称。
在miRWalk中,用户可以检索基因和miRNA信息,同时支持一次性检索多个对象。数据库提供筛选功能,允许用户根据多个预测程序的结果来选择目标。网页界面支持基于结合位置和评分值的筛选,高评分表示可能性高。用户可以将筛选结果导出为Excel文件,或通过点击链接查看预测靶基因汇总条目。miRWalk还提供调控网络可视化功能和基因集富集分析(GSEA),图形化显示miRNA和基因之间的关系,支持导出为多种格式。
对于大规模数据预测,miRWalk提供数据库下载选项,用户可将整个数据库下载至本地进行批处理分析。下载文件通常包含基因的5UTR、CDS和3UTR区域的miRNA预测信息,重点在3UTR区域。下载后的数据需要通过脚本进行处理和筛选,以提取和分析预测结果。miRWalk不仅提供了基础功能,还鼓励用户探索更多分析方法和应用场景。
三、资源|基础研究常用数据库汇总
分子生物学是研究生命现象本质的科学,其基础是中心法则,涉及到基因信息、基因表达、基因功能分析、转录因子、miRNA分析与预测等多个方面。为支持分子生物学研究的发展,强大数据库的构建至关重要。
基因信息的查询主要依赖于三大数据库:GeneCards、BioGPS和UCSC Genome Browser。GeneCards提供全面的人类基因信息,整合了约150个在线基因中心数据,包括基因组、转录子、别名、结构域、药物、表达和定位等信息。BioGPS是一个自由拓展和定制的基因注释网站,主要包含人类、小鼠和大鼠的基因。UCSC Genome Browser是一个交互式基因组浏览器,提供了多种脊椎动物、无脊椎动物和主要模式生物的基因组序列数据。
基因表达信息可通过Gene Expression Omnibus(GEO)、ArrayExpress和The Cancer Genome Atlas(TCGA)获取。GEO是一个公共功能基因组数据库,支持符合MIAME标准的数据提交,并提供工具帮助用户查询和下载实验并设计基因表达谱。ArrayExpress归档芯片和测序平台的功能基因组学数据,以支持可重复的研究。TCGA网站绘制出33种癌症关键基因组变化的综合多维图谱,旨在应用高通量基因组分析技术,帮助人们更好地了解癌症。
基因功能分析可以通过DAVID、Metascape和XTalkDB等工具进行。DAVID提供全面的功能注释工具,能够确定生物学主题、发现功能相关基因组、群集冗余注释术语、在BioCarta和KEGG信号通路图上实现基因可视化,并在二维视图上显示相关的多个基因。Metascape是一个免费的基因注释和分析资源库,旨在帮助生物学家理解一组正交蛋白发现研究中的常见或独特途径和蛋白质网络。XTalkDB研究信号通路间相互作用,为系统生物学研究提供重要支持。
转录因子的研究主要依赖于iRegulon、TFcheckpoint等数据库。iRegulon包含转录因子及其直接转录DNA序列组成的数据库,并提供插件识别调节子。TFcheckpoint则是人类、小鼠和大鼠转录因子数据库,手动检索可以获得数据。
miRNA分析与预测的数据库有starBase、miRTarBase、miRWalk、miRBase、TargetScan和DIANA。starBase研究miRNA-ncRNA、miRNA-mRNA、ncRNA-RNA、RNA-RNA、RBP-ncRNA和RBP-mRNA相互作用。miRTarBase是经过实验验证的microRNA-target相互作用数据库。miRWalk提供已验证和预测的miRNA结合位点信息。miRBase是已发表的miRNA序列和注释的数据库。TargetScan通过搜索miRNA的种子区域匹配的保守位点预测miRNA的靶点。DIANA是miRNA研究工具的集锦,提供microT-CDS预测、LncBase v.2、miRGen v.3和Mirpub等功能。
除了基因相关数据库,L1000FWD和MalaCards两个数据库也值得关注。L1000FWD提供超过16,000种药物和小分子诱导的基因表达特征,可实现直观化与可视化的功能。MalaCards是人类疾病综合性数据库,整合了专业和一般疾病信息。